Gérez vos données non équilibrées sous Python
Oversampling, SMOTE, undersampling, ou encore un mixte de toutes ces techniques : le rééchantillonnage pour équilibrer une classe minoritaire
Evaluation des données
La question des données déséquilibrées se pose souvent dans le cadre d'une problématique de classification. Une ou plusieurs classes sont minoritaires par rapport à d'autres. Bien qu'il s'agisse en soi d'une information importante, un déséquilibre trop prononcé peut nuire à la pertinence d'un modèle.
Voici, ci-dessous un exemple :
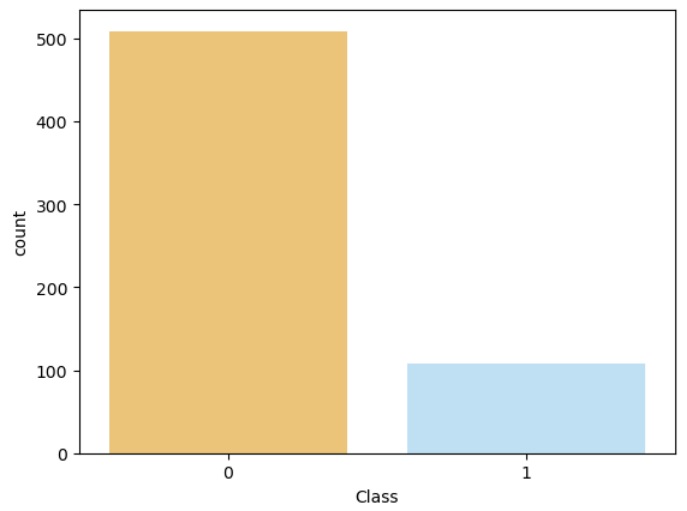
Comme vous pouvez le constater ci-dessus, nous sommes en présence d'une problématique liée à une classification binaire. La classe 0 est majoritaire, la classe 1 minoritaire. Cette dernière ne représente pas plus de 20% de la totalité des observations. Il est courant de qualifier un déséquilibre de léger si la classe minoritaire représente 20 à 40% des observations, de moyen si le taux est inferieure à 20% et enfin de fort si celui-ci est inferieur à 1%.
Plusieurs techniques de rééchantillonnage existent. Dans le cadre de cet article, nous allons évoquer le random oversampling, le SMOTE, le random undersampling et enfin nous verrons comment mixer ces différentes techniques via un pipeline.
Random oversampling
La technique du random oversampling est relativement basique puisqu'elle va tout simplement consister à augmenter les effectifs de la classe minoritaire en dupliquant des observations choisies aléatoirement.
Déroulons un petit exemple pour s'en convaincre. Nous allons tout d'abord vérifier si notre dataset comporte des observations en doublons. Notons qu'il s'agit d'un dataset quelconque pour lequel nous n'avons pas, dans cet article, traité du chargement. Il est juste nécessaire de savoir que les facteurs sont regroupés dans un dataframe x_train, la classe cible est isolée dans un dataframe y_train.
import numpy as np
import pandas as pd
#Nous convertissons ici nos dataframe en tableau numpy en vue du traitement a venir
x_train = x_train.to_numpy()
y_train = y_train.to_numpy()
#Nous determinons le nombre d'observations dupliquees
pd.DataFrame(x_train).duplicated().sum()
0
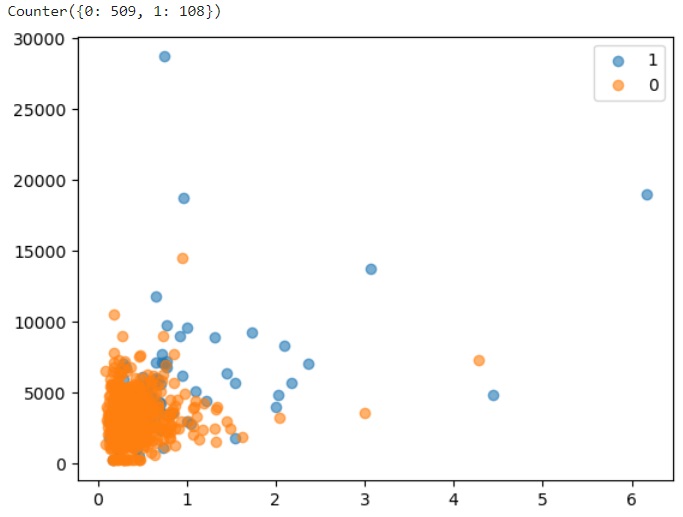
Comme nous pouvons le constater, le dataset original ne comporte pas d'observations en doublons. Dressons à présent un nuage de points nous permettant de comparer visuellement nos 2 classes binaires. Pour cela nous allons nous baser arbitrairement sur les 2 premières colonnes de notre dataset.
from collections import Counter
from matplotlib import pyplot
from numpy import where
counter = Counter(y_train)
print(counter)
for label, _ in counter.items():
row_ix = where(y_train == label)[0]
pyplot.scatter(x_train[row_ix, 0], x_train[row_ix, 1], label=str(label), alpha=0.6)
pyplot.legend()
pyplot.show()
Appliquons un random oversampling sur nos données. Disons que nous voulons par exemple que notre classe minoritaire soit représentée à hauteur de 80% des effectifs de la classe majoritaire :
from imblearn.over_sampling import RandomOverSampler
over = RandomOverSampler(sampling_strategy=0.8)
x_train, y_train = over.fit_resample(x_train, y_train)
counter = Counter(y_train)
print(counter)
for label, _ in counter.items():
row_ix = where(y_train == label)[0]
pyplot.scatter(x_train[row_ix, 0], x_train[row_ix, 1], label=str(label), alpha=0.6)
pyplot.legend()
pyplot.show()
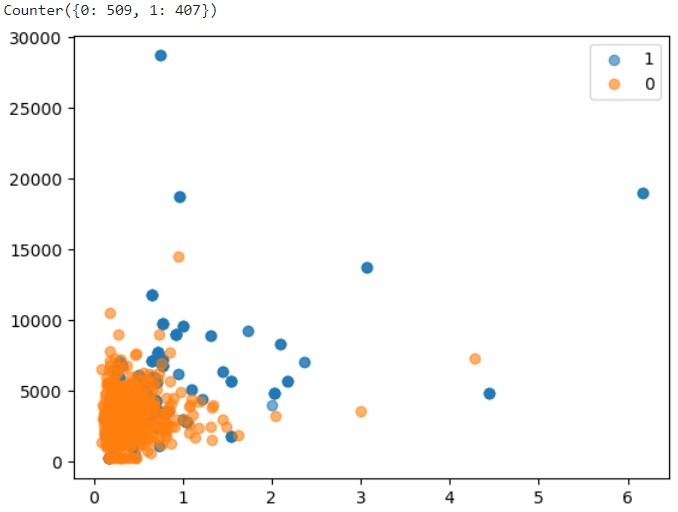
Nous constatons que les effectifs de la classe 0, majoritaire, n'ont pas changé. Ceux de la classe minoritaire 1, par contre, sont passés de 108 à 407. Le nuage de points n'a pas évolué, les observations bleues, associées à la classe 1 sont en apparence les mêmes. Nous pouvons cependant noter qu'ils sont plus « foncés » que sur le nuage précèdent. L'indice de transparence alpha que nous avons spécifié sur le nuage nous permet en effet de voir que les points se sont superposés les uns sur les autres. Pour cause, puisque l'oversampling s'est contenté de dupliquer nos observations. Ainsi, si nous demandons à nouveau le nombre d'observations en doublons, nous obtenons ceci :
#Nous determinons a nouveau le nombre d'observations dupliquees
pd.DataFrame(x_train).duplicated().sum()
299
En matière d'oversampling, il existe une méthode plus sophistiquée : SMOTE, dont nous allons parler dans le point suivant.
SMOTE
SMOTE signifie Synthetic Minority Oversampling Technique. Cette méthode est également une technique d'oversampling. Son objectif est, par conséquent, d'augmenter le nombre d'observations associées à la classe minoritaire. A la différence de la méthode précédemment décrite, SMOTE va tout d'abord choisir de façon aléatoire des observations issues de la classe minoritaire puis choisir, également de façon aléatoire k plus proches voisins afin de synthétiser une nouvelle observation.
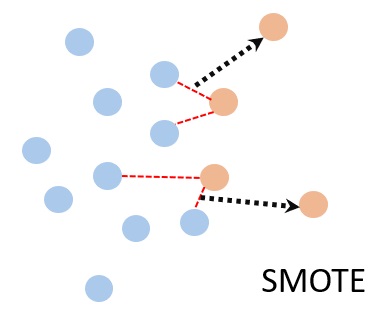
Appliquons SMOTE à nos données, en lieu et place de l'oversampling précèdent, puis dressons le nuage de points :
from imblearn.over_sampling import SMOTE
over = SMOTE(sampling_strategy=0.8)
x_train, y_train = over.fit_resample(x_train, y_train)
counter = Counter(y_train)
print(counter)
for label, _ in counter.items():
row_ix = where(y_train == label)[0]
pyplot.scatter(x_train[row_ix, 0], x_train[row_ix, 1], label=str(label), alpha=0.6)
pyplot.legend()
pyplot.show()
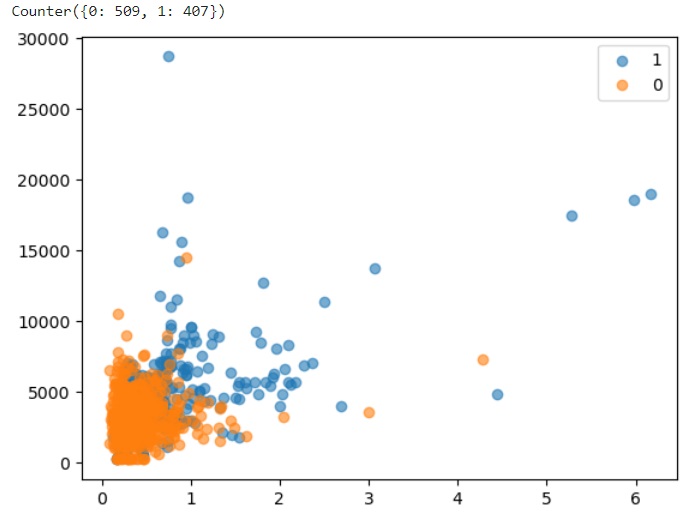
Nous avons appliqué la même stratégie d'échantillonnage de 80%, nous avons par conséquent le même effectif au final qu'avec la méthode précédente. Nous pouvons cependant noter que la classe minoritaire laisse apparaitre sur le nuage plus de points distincts. Ceci est dû à la méthode décrite plus haut. Voyons si des doublons sont apparus dans le set de données :
#Nous determinons a nouveau le nombre d'observations dupliquees apres SMOTE
pd.DataFrame(x_train).duplicated().sum()
0
Comme prévu, aucun doublon n'a été détecté, SMOTE a donc bien créé de nouvelles observations.
Random undersampling
La technique de random undersampling consiste à diminuer, au contraire, les effectifs de la classe majoritaire, en supprimant des observations, associées à cette classe, de façon aléatoire.
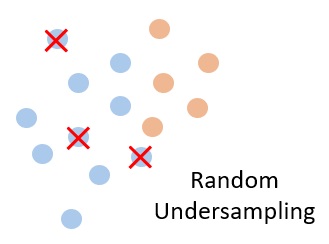
Appliquons un random undersampling à nos données originales puis dressons le nuage de points associé :
from imblearn.under_sampling import RandomUnderSampler
under = RandomUnderSampler(sampling_strategy=0.8)
x_train, y_train = under.fit_resample(x_train, y_train)
counter = Counter(y_train)
print(counter)
for label, _ in counter.items():
row_ix = where(y_train == label)[0]
pyplot.scatter(x_train[row_ix, 0], x_train[row_ix, 1], label=str(label), alpha=0.6)
pyplot.legend()
pyplot.show()
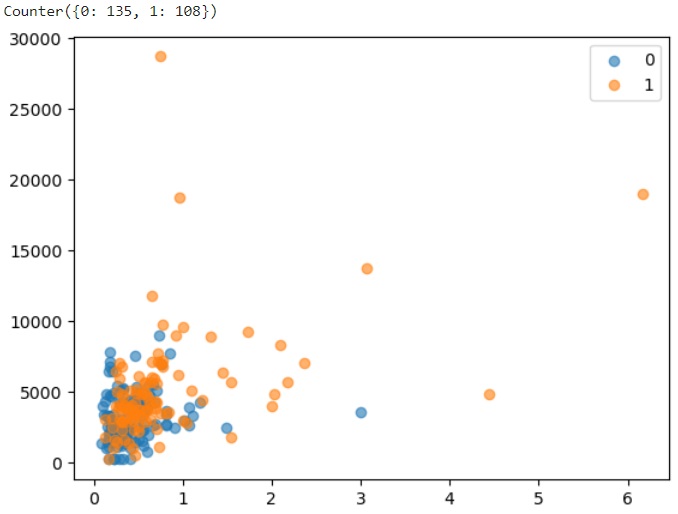
Nous avons conservé la stratégie d'échantillonnage à 80%, ce qui signifie que le random undersampling va diminuer les effectifs de la classe majoritaire jusqu'à ce que ces derniers supérieurs de 20% à la classe minoritaire. Nous pouvons le constater sur le rendu ci-dessus puisque nous avons désormais 135 observations associées à la classe 0 et 108 associées à la classe 1. Le nuage s'en trouve par conséquent plus aéré.
Pipeline
Pour finir, nous allons mixer la technique SMOTE avec celle de l'undersampling via un pipeline afin d'appliquer successivement sur nos données ces deux méthodes. En terme de stratégie d'échantillonnage, nous allons opter pour un taux de 40% sur le SMOTE et de 50% sur l'undersampling :
from imblearn.over_sampling import SMOTE
from imblearn.under_sampling import RandomUnderSampler
from imblearn.pipeline import Pipeline
# define pipeline
over = SMOTE(sampling_strategy=0.4)
under = RandomUnderSampler(sampling_strategy=0.5)
steps = [('o', over), ('u', under)]
pipeline = Pipeline(steps=steps)
x_train, y_train = pipeline.fit_resample(x_train, y_train)
counter = Counter(y_train)
print(counter)
for label, _ in counter.items():
row_ix = where(y_train == label)[0]
pyplot.scatter(x_train[row_ix, 0], x_train[row_ix, 1], label=str(label), alpha=0.6)
pyplot.legend()
pyplot.show()
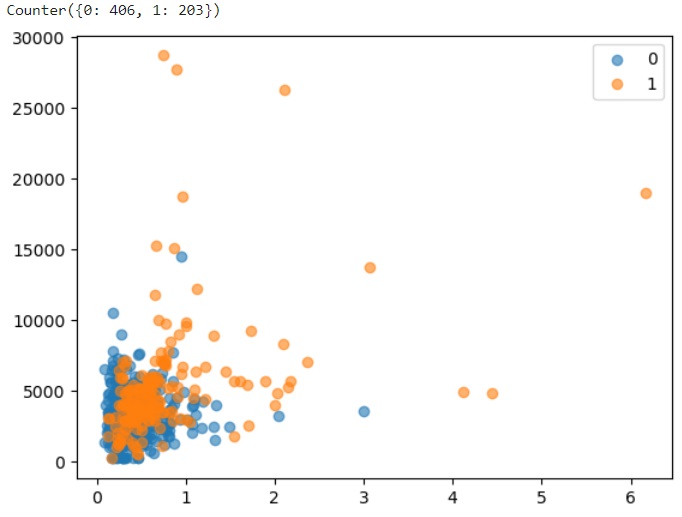
Le SMOTE appliqué ci-dessus a fait passer notre classe minoritaire de 108 observations à 203 (soit 40% de 509, l'effectif de la classe majoritaire). L'undersampling qui a suivi a coupé dans les effectifs de la classe majoritaire pour les faire passer de 509 à 406 (soit 50% de plus que les effectifs de la classe minoritaire).
Crédits
Miniature issue de vecstock sur Freepik
Retrouvez dans la rubrique "Nos datasets" toutes les données dont vous aurez besoin pour tester et pratiquer !

